고감도 Whole Genome Amplification for Low-Input DNA
몇 femtograms(fg) 수준의 DNA로 전체 게놈을 증폭할 수 있다는 사실이 믿기시나요? 여러분의 소중한 single-cell 샘플, 희귀 미생물, 혹은 고대 DNA를 다룰 때, Whole Genome Amplification(WGA)만이 downstream 분자 분석을 가능하게 합니다. 이 가이드에서는 NEB의 phi29-XT WGA Kit를 깊이 파헤치고, 핵심 기능, 최적 워크플로우, 그리고 debranching과 RNA 제거에 대한 실용적인 팁을 알려드립니다. 이 글을 읽고 나면, 단 10 fg의 입력 DNA로도 견고하고 고수율의 게놈 라이브러리를 생성할수 있을 것입니다.
유전체학에서 WGA가 중요한 이유
Whole Genome Amplification은 시작 DNA 양이 너무 적어 표준 라이브러리 준비가 어려운 경우에도 전체 게놈을 균일하게 증폭할 수 있게 해줍니다. 특히 다음과 같은 경우에 필수적입니다:
- Single-cell sequencing: 세포 하나당 게놈 DNA가 피코그램(picogram) 단위라 균일한 증폭이 필수적입니다.
- Forensic investigations: 범죄 현장에서 얻은 미량 DNA는 downstream 분석을 위해 증폭이 필요합니다.
- Microbiome profiling: 개별 미생물 세포나 저빈도 종의 DNA를 분리할 때 WGA가 샘플 손실을 방지합니다.
- Ancient DNA studies: DNA가 극도로 적거나 훼손된 샘플은 효율적인 증폭으로만 복구할 수 있습니다.
여러 WGA 방법 중 Multiple Displacement Amplification(MDA)는 High fidelity phi29 DNA polymerase를 사용하여 넓은 게놈 커버리지, 최소한의 바이어스, 높은 수율을 제공하므로 표준으로 자리 잡았습니다. 그리고 바로 phi29-XT WGA Kit가 그 핵심 솔루션입니다.
phi29-XT WGA Kit의 주요 특징
phi29-XT WGA Kit (NEB #E1604S/L)는 whole genome amplification을 간소화하고 가속화하도록 설계된 키트로, 뛰어난 감도를 자랑합니다. 주요 특징은 다음과 같습니다.
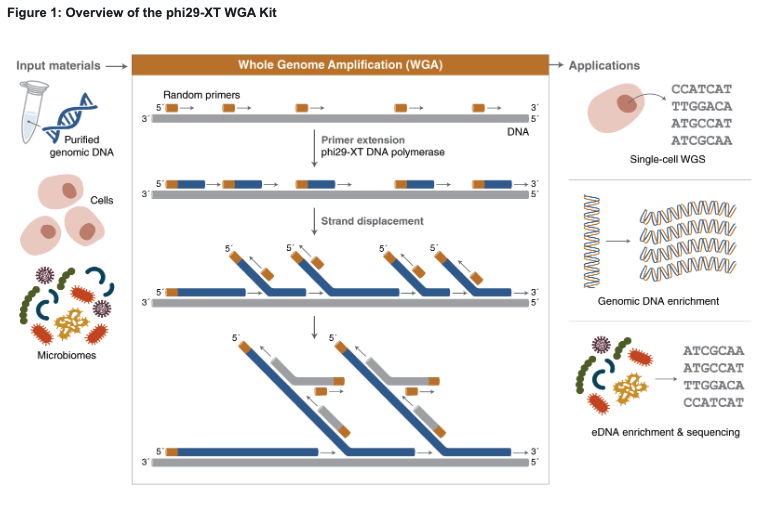
- 향상된 phi29-XT DNA Polymerase
키트에는 phi29 DNA polymerase의 엔지니어링 변종인 phi29-XT가 포함되어 있으며, thermostability와 민감도가 크게 개선되었습니다. 이로 인해 고온에서도 활성이 유지되어 비특이적 증폭이 줄고, 더 균일한 커버리지를 얻을 수 있습니다. - 초고감도: 10 fg 수준의 DNA부터 증폭
세균 하나에 해당하는 DNA 양 또는 그 이하에서도 WGA 반응이 가능합니다. phi29-XT polymerase는 최소 10 femtograms(fg) 수준의 입력량을 안정적으로 증폭할 수 있어 single-cell이나 rare-cell 유전체학에 최적화되어 있습니다. - 2시간의 간단한 워크플로우
전체 WGA 반응은 denaturation부터 inactivation까지 2시간 미만에 완료됩니다. 상온에서 5분간 denaturation, 42°C에서 90분간 isothermal 증폭, 그리고 65°C에서 10분간 열불활성화를 통해 마이크로그램(µg) 단위의 산물을 신속히 얻을 수 있습니다. - 인간 및 미생물 DNA에 대한 전체 게놈 표현성
random primers와 strand-displacement 활성으로 인해 거의 전체 게놈을 균일하게 증폭할 수 있어 downstream NGS, array 분석, genotyping 등에 이상적입니다. - 알칼리성 조건에서의 잔존 RNA 제거
샘플에는 종종 잔류 RNA가 있어 downstream 프로토콜에 방해가 됩니다. 키트의 알칼리성 denaturation 단계에서 RNA가 효과적으로 분해(digestion)되어 순수한 DNA 템플릿만 남게 됩니다. 추가적인 RNase 처리가 필요 없습니다. - 다양한 downstream 애플리케이션 호환
single-cell WGS, metagenomic sequencing, copy-number variation 분석 등 다양한 용도에 그대로 사용할 수 있습니다. 단, debranching 과정만 거치면 됩니다 (아래 단계 참고).
단계별 워크플로우
아래는 phi29-XT WGA Kit를 이용한 전형적인 워크플로우입니다. NGS나 기타 고해상도 애플리케이션에 사용할 샘플의 경우 debranching 단계를 반드시 확인하세요.
자세한 매뉴얼은 phi29-XT WGA E1604 Manual (neb.com) 에서 확인하세요.

1. 샘플 준비 및 Denaturation
- DNA 정량: Qubit과 같은 고감도 형광 정량법으로 최소 10 fg DNA가 있는지 확인합니다.
- 반응 버퍼 및 랜덤 프라이머 혼합: 제공된 phi29-XT reaction buffer와 random primers에 템플릿 DNA를 넣고 혼합합니다.
- 상온에서 Denature(5분): 알칼리성 denaturation으로 이중가닥이 풀리는 동시에 잔여 RNA가 분해됩니다. 5분 후 즉시 중화합니다.
팁: Denaturation 동안 샘플을 빠르게 취급하여 긴 게놈 조각이 물리적으로 손상되지 않도록 합니다.
2. Whole Genome Amplification (42°C에서 90분)
- phi29-XT Polymerase 첨가: Denature된 템플릿+프라이머 혼합물에 효소를 넣습니다.
- 42°C에서 인큐베이션: 폴리머레이스가 프라이머를 신장시키고 기존 가닥을 displacement하며, multiple displacement가 일어나 긴 브랜치 형태의 DNA 산물이 형성됩니다.
- 반응 시간 조절: 표준은 90분이지만, 60–120분 사이에서 원하는 수율에 맞춰 조절할 수 있습니다. 반응 시간을 늘리면 더 많은 DNA를 얻을 수 있으나 약간의 바이어스가 생길 수 있습니다.
팁: 반응 중 온도가 일정하게 유지되도록 주의하세요. 온도 변동은 증폭 균일성을 떨어뜨립니다.
3. 열불활성화(65°C에서 10분)
- 효소 활성을 종결: 65°C에서 10분간 가열하여 phi29-XT polymerase를 완전히 비활성화합니다.
- 얼음 위로 냉각: 반응을 4°C로 즉시 내려 비특이적 배경 활성을 방지합니다.
이 시점에서 10 fg의 샘플에서 5–10 µg의 증폭 DNA를 얻을 수 있습니다. 다만 이 산물은 branched 구조이므로, blunt-end 라이게이션이나 시퀀싱 클러스터 생성 시 문제를 일으킬 수 있습니다. 따라서 다음 단계인 debranching이 필요합니다.
WGA product Debranching
phi29 기반 WGA는 branched DNA 구조물을 생성하며, 이 구조는 다음과 같은 문제를 유발할 수 있습니다.
어플리케이션에 따라 purification 없이 사용할 수 있지만 NGS를 진행하신다면 debranching과정을 거치시기 바랍니다.
- 라이게이션 효율 저하
- 어댑터 부착 어려움
- 시퀀싱 플랫폼에서의 클러스터 형성 불안정
이 브랜치 구조를 “평탄화(flatten)”하기 위해 다음 절차를 수행하세요.
1. 필요한 시약 준비
- NEBuffer 2 (NEB): T7 Endonuclease I의 최적 활성을 보장합니다.
- T7 Endonuclease I (NEB): 브랜치/교차 모양 구조를 인식하여 절단하는 효소입니다.
2. Debranching 프로토콜
- 반응 설정: WGA 산물을 1× NEBuffer 2와 혼합합니다.
- T7 Endonuclease I 첨가: WGA 산물 1 µg당 5–10 units를 사용합니다.
- 37°C에서 30–45분 인큐베이션: 효소가 브랜치 지점을 절단하여 주로 linear, high-molecular-weight 조각을 생성합니다.
- 65°C에서 10분간 열불활성화: T7 Endonuclease I 활성을 중단해 과다 절단을 방지합니다.
- DNA 정제: Ampure XP 비드 정제 또는 칼럼 기반 정제법으로 효소와 버퍼 잔류물 제거합니다.
3. 품질 확인
- Pulse-Field Gel 또는 TapeStation: Debranched 산물이 10–50 kb 사이의 스미어(smeared band)로 나타나고 고분자량이 남아 있지 않은지 확인합니다.
- Qubit으로 정량: 이후 라이브러리 준비를 위해 농도를 정확히 측정합니다.
알칼리성 RNA 제거의 장점
많은 WGA 프로토콜에서는 RNA 오염이 문제입니다. RNA가 있으면,
- 정량 시(예: 스펙트로포토미터) 배경 노이즈가 증가합니다.
- 어댑터 라이게이션이나 end-repair 효소 효율이 떨어집니다.
- downstream PCR 기반 분석에서 프라이머 결합을 방해할 수 있습니다.
phi29-XT WGA Kit는 첫 단계의 알칼리성 denaturation을 통해 이 문제를 해결합니다. 고온(혹은 알칼리성) 조건에서,
- RNA 가수분해(RNA Hydrolysis): 높은 pH 환경이 RNA의 인산다이에스터 결합을 파괴합니다.
- DNase 비활성화: 중화 단계 후 온전한 고품질 DNA만 반응에 남습니다.
- 추가 RNase 불필요: 별도의 RNase 처리 없이도 RNA를 제거할 수 있어 시간과 샘플을 절약합니다.
실용 팁 & 문제 해결
최고의 WGA 키트도 때로는 난관에 부딪힐 수 있습니다. 일관성 있고 고품질의 증폭을 위해 다음 best practices를 따르세요:
- 초고순도 DNA 사용: 남아 있는 페놀, 에탄올 등 저해제가 있으면 phi29-XT polymerase 활성이 떨어집니다. 고순도 물 또는 TE 버퍼를 사용하세요.
- 피펫팅 단계 최소화: 긴 DNA는 쉽게 물리적으로 절단될 수 있습니다. 와이드 보어 팁 또는 팁 끝을 약간 잘라 사용하는 것이 도움이 됩니다.
- 신선한 시약 사용: phi29-XT polymerase는 –20°C에 보관하고, 잦은 동결-해동을 피하세요.
- 음성 대조(Negative Control) 포함: NTC (no-template control)를 항상 포함해 환경적 오염 여부를 확인하세요. 특히 초저 입력(Ultra-Low Input) 작업 시 중요합니다.
- 반응 시간 최적화: 배경 증폭이 높거나 커버리지 균일도가 낮다면 증폭 시간을 60분으로 단축해 보세요.
- Debranching 확인: T7 Endonuclease I 처리 후 소량을 겔에 올려 제대로 debranching이 되었는지 항상 확인하세요.
애플리케이션 & Downstream 워크플로우
Debranched된 고분자량 DNA를 확보한 후에는 다양한 downstream 애플리케이션이 가능합니다:
1. NGS 라이브러리 준비
- Illumina: PCR-free 또는 PCR-enriched 라이브러리 준비에 NEBNext Ultra II 키트를 사용하세요.
- Oxford Nanopore: 길이가 10–50 kb 수준인 debranched 조각은 long-read sequencing에 이상적입니다.
- PacBio: >15 kb 삽입(fragment)을 사이즈 선택하면 높은 정확도를 얻을 수 있습니다.
2. Copy-Number Variation (CNV) 분석
균일한 전체 게놈 표현성 덕분에 구조 변이(structural variants), 결실(deletions), 중복(duplications) 등을 정확하게 탐지할 수 있습니다.
3. Metagenomic Profiling
환경 시료나 미생물 분리주(isolate)에서도 단 single-cell 수준으로 박테리아나 바이러스 전체 게놈을 캡처할 수 있습니다.
4. Ancient DNA & Forensics
훼손되거나 매우 적은 양의 고대 DNA를 WGA로 복구해, 게놈을 재구성할 수 있습니다.
자주 묻는 질문 (FAQ)
결론
NEB의 phi29-XT WGA Kit는 초저 입력(Ultra-Low Input) DNA를 빠르고 안정적으로 증폭해야 하는 연구자들에게 최적의 솔루션입니다. 엔지니어링된 polymerase(phi29-XT), 알칼리성 RNA 제거, 간소화된 프로토콜이 결합되어 단 2시간 이내에 고품질의 debranched 게놈 DNA를 마이크로그램(µg) 단위로 얻을 수 있습니다. Single-cell genomics, 미생물 다양성 연구, 법과학(Forensic) 분석 등 어떤 분야에서도, 이 키트는 귀중한 DNA 샘플의 잠재력을 최대한 끌어낼 수 있도록 도와줄 것입니다.
'신기한_바이오 > NGS' 카테고리의 다른 글
| Mastering Human and Bacterial rRNA Depletion with NEBNext Kits (0) | 2025.06.05 |
|---|